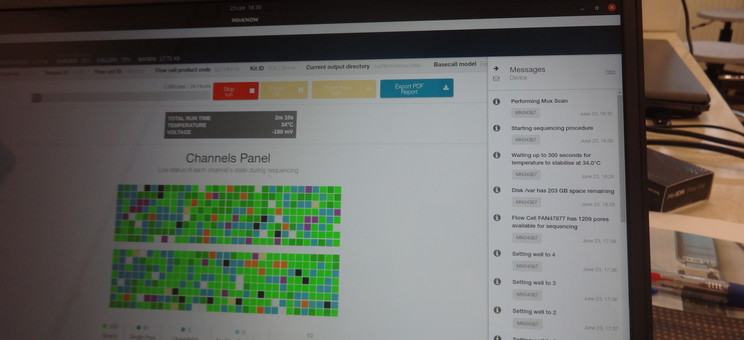
W dniach 22-26.06.2020, w ramach realizacji projektu „Technologia Oxford Nanopore: optymalizacja enzymów oraz analizy danych genomicznych pod kątem zastosowań komercyjnych” został przeprowadzony pierwszy eksperyment sekwencjonowania DNA z wykorzystaniem technologii Oxford Nanopore. Eksperyment podzielono na dwie części – doświadczalną realizowaną w Zakładzie Biotechnologii i Bioinformatyki Wydziału Chemicznego oraz bioinformatyczną wykonaną w Zakładzie Systemów Złożonych Wydziału Elektrotechniki i Informatyki. Sprawne przeprowadzenie sekwencjonowania, jak również prawidłową analizę uzyskanych danych, nadzorował mgr Jan Gawor z Pracowni Sekwencjonowania DNA i Syntezy Oligonukleotydów Instytutu Biochemii i Biofizyki PAN. W realizację eksperymentu byli zaangażowani m.in.: dr Marta Sochacka-Piętal, dr Michał Pietal, mgr Małgorzata Semik, mgr Michał Wroński, dr inż. Dominik Strzałka, mgr Magdalena Totoń, inż. Michał Ćmil i mgr inż. Anna Czmil. Niezbędnej pomocy organizacyjno-technicznej udzielił także dr hab. inż. Mirosław Tyrka, prof. PRz. Materiałem wyjściowym w części doświadczalnej było DNA z termotolerancyjnych oraz termofilnych szczepów bakterii z rodzaju Bacillus. W trakcie części bioinformatycznej eksperymentu wykonawcy projektu zapoznali się z szeregiem narzędzi bioinformatycznych (m.in. Bandage, Guppy, NanoPlot, Fly, Canu), wykorzystywanych w składaniu de novo genomów bakteryjnych. W wyniku przeprowadzonego eksperymentu uzyskano de novo kompletne sekwencje genomowe analizowanych szczepów Bacillus sp.
Więcej na stronie: https://zsz.prz.edu.pl/dzialalnosc-naukowa/projekty/technologia-oxford-nanopore/aktualnosci